Mécanismes et facteurs de virulence
Nouveaux facteurs de virulence & Dickeya secretome
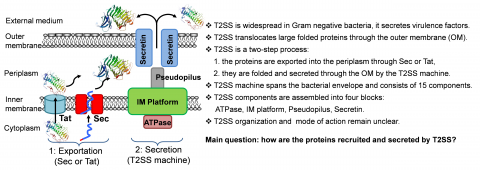
Les sécrétomes T2SS (système de sécrétion de type 2) récents de certaines protéobactéries pathogènes ont révélé un large spectre d'exoprotéines fonctionnellement différentes, impliquées dans la virulence. Pourtant, l'ensemble des exoprotéines sécrétées par le T2SS de Dickeya reste à identifier et notre étude récente (Liu et al. 2019) a montré que ces protéines pourraient avoir un large spectre d'action. Pour définir l'ensemble des substrats T2SS, on comparera le sécrétome des souches de type sauvage et ses mutants non sécréteurs cultivés dans des conditions imitant celles des plantes. Cette analyse se concentrera sur différentes espèces de Dickeya, dont D. solani, un important pathogène des plantes émergent en Europe. Cela nous permettra de définir le "core T2SS-secretome" et le secrétome indépendant du T2SS", dans le genre Dickeya. Alors que le sécrétome central contient les fonctions générales nécessaires à la pourriture molle des plantes, les substrats T2SS accessoires ou uniques pourraient être acquis plus récemment par Dickeya pour conquérir de nouvelles niches ou hôtes. L'analyse quantitative du protéome sera effectuée dans l'installation de MS de l'IBCP (Lyon). Dans une autre approche plus directe, les nouvelles protéines T2SS sécrétées sont systématiquement recherchées parmi les protéines fortement induites dans les infections végétales (données RNAseq). Les protéines de fonction inconnue portant le peptide signal (obligatoire pour le T2SS) sont une cible privilégiée pour une telle analyse. La fonction, la régulation et le rôle dans la virulence de ces nouveaux substrats T2SS seront étudiés, ainsi que leur présence dans d'autres pathogènes végétaux. Une nouvelle protéine IbpS porteuse du fer a récemment été identifiée par cette approche (Liu et al., 2019).
Définir un coeur commun de facteurs de colonisation de l’hôte (végétal, insecte)
Nos études récentes ont montré une large présence de diverses espèces de Dickeya dans divers milieux tels que le sol, les plantes saines ou l'eau (Hugouvieux-Cotte-Pattat et al, 2019). Ces bactéries représentent une source potentielle d’émergence de maladies, mais on ne sait pas comment les réservoirs environnementaux influent sur leur survie, leur multiplication et leur pathogénie. Pour étudier les déterminants génétiques du mode de vie bactérien tant dans les plantes, dans l'eau douce ou les insectes, vecteurs majeurs de dissémination des pathogènes à travers les agroécosystèmes, nous utilisons le Tn-Seq, une mutagenèse innovante associée à une technique de séquençage à haut débit, récemment mise en œuvre dans le laboratoire (Royet et al., 2019). Cette étude comparative (projet ANR TNPHYTO) permettra de définir un socle commun de facteurs essentiels à la colonisation végétale et spécifiques à certaines espèces.
L’insecte est un nouvel hôte (milieu/vecteur) étudié dans l’équipe au travers des interactions bactériennes précoces établies par les bactéries* avec leurs cuticules (cf EPI-Cuticle).
*eg le double pathogène Dickeya dadantii ou le double symbiote Serratia symbiotica